今回は,離散フーリエ変換でえられるギザギザなパワースペクトルを,滑らかにする「平滑化」についてです.いろいろ書きたいことはありますが,まずは,Rでお世話になることが多い"spectrum"というコマンドのオプション"spans"について説明します.今回の説明は,私が数値実験をして確認したことに基づいていますので,間違いがあるかもしれません.他人を簡単に信じないでください.
1. パワースペクトルの平滑化
一般的な平滑化の説明をしておきます.離散フーリエ変換を使って計算されたパワースペクトルの平滑化は,重み
(
)を使って,
と表されます.
移動平均で平滑化する場合は,
(
)
です (下図).時系列の移動平均と同じように,パワースペクトルが平滑化されます.

2. "spans"の使用例
Rの"spectrum"コマンドを使ったことがない人もいるかもしれませんし,使ったことはあっても,"spans"オプションを指定したことがない人もいるかもしれませんので,まずは,数値実験の結果をお見せします.時系列は2次自己回帰過程 (,
)で生成しました.時系列のベクトルデータをx.simとすれば,使ったコマンドは,
spectrum(x.sim, ...)
です."..."の部分はオプションコマンドが入ります.例えば,グラフを描かない場合は,
spectrum(x.sim, plot=FALSE)
とします.
"spans"を使った結果は下の図です.平滑化していない結果 (左上)と比べて,平滑化すると,解析的結果 (赤破線)と推定結果 (青実線)の一致が良くなっています.
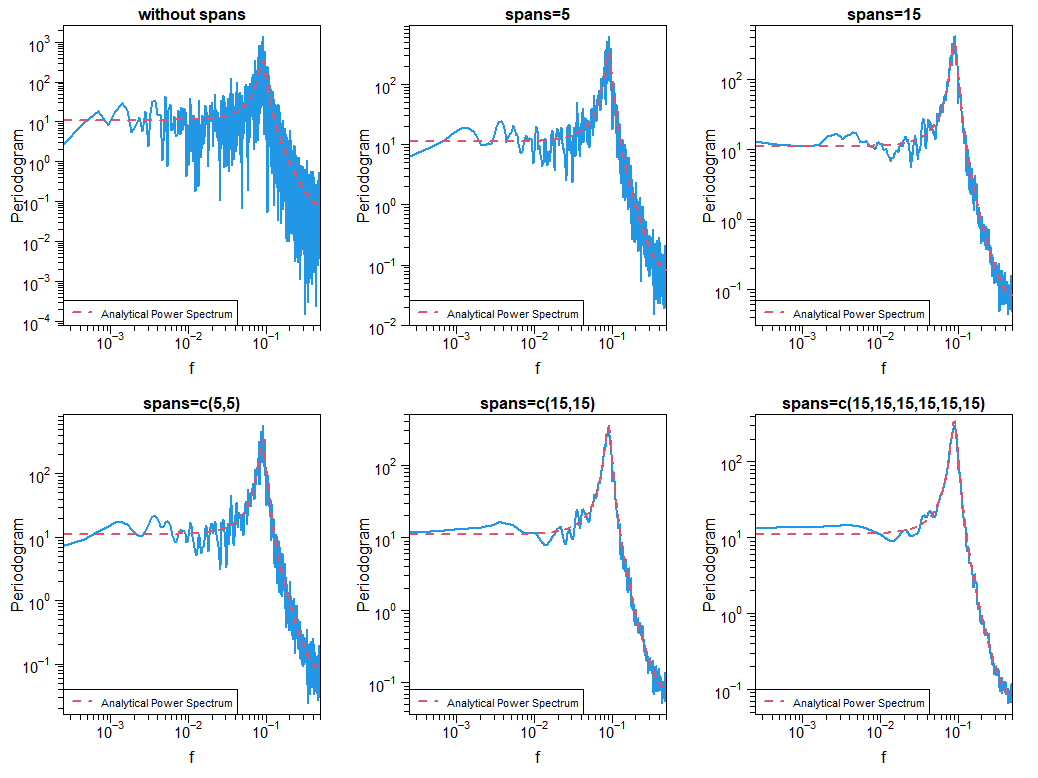
指定した"spans"は,各図の上のタイトル部分に書いてあります.図の左上から順番に,対応するRのコマンドを書けば,
tmp <- spectrum(x.sim, plot=FALSE) tmp <- spectrum(x.sim, spans=5, plot=FALSE) tmp <- spectrum(x.sim, spans=15, plot=FALSE) tmp <- spectrum(x.sim, spans=c(5,5), plot=FALSE) tmp <- spectrum(x.sim, spans=c(15,15), plot=FALSE) tmp <- spectrum(x.sim, spans=c(15,15,15,15,15,15), plot=FALSE)
です.この場合,"tmp$freq"に周波数,"tmp$spec"に推定されたスペクトル密度が入っています.
"spans"を指定すれば,平滑化できていることは見て取れます.でも,これで何をやっているの? "spans=c(15,15,15,15,15,15)"って,マニュアルの使用例に載っていないし,ふざけてんのか? と感じる人も多いと思います.
3. Rのマニュアルの説明を見ると
"spectrum"を実行した場合,デフォルトで実際の動いているのは"spec.pgram"のようです.Rコンソールで
?spec.pgram
を実行すれば,マニュアルを見ることができます.そこでは,"spans"のオプションについて,
「ピリオドグラムを平滑化するために使用される修正ダニエルスムーザ (modified Daniell smoothers)の幅を与える奇数を並べたベクトル.」
と書いてあります.つまり,"spans=c(3,5,15,9,7,11)"のように,奇数のベクトルを指定しろということです.
以下で,"spans=c(...)"の意味を読み解いていきます.
4. 修正ダニエルスムーザ (modified Daniell smoothers)とは
まず,修正版ではない,元の「ダニエルスムーザ」は,単なる移動平均フィルタ (スムーザ)です.つまり,最初に説明した例と全く同じです.
平均をとる全幅 (点数)が
ダニエルスムーザと修正ダニエルスムーザの違いは,両端の点にあります (下図).

上図の右のように修正ダニエルスムーザでは,両端の重みが,中央部の重みの半分になります.つまり,式で書くと以下です.
平均をとる全幅 (点数)が
何で端の重みのみ半分にするのか,私には理由は分かりません.想像で考えると,のときなど,幅が小さいときに,中央の点の重みを大きくできるので,元の構造を壊さないように軽~くスムージングするには修正版が良いかもしれません.
が大きくなると,ダニエルも修正ダニエルも,結果はほぼ同じだと思います.
5. "spans"で指定する値の意味
"spectrum"コマンドのオプション"spans"では,修正ダニエルスムーザの長さを指定しています (
).以下のように,
spectrum( ... , spans=L, ... )
1つの値だけ指定すれば,上で説明した修正ダニエルで平滑化されます.
さらに,以下のように,
spectrum( ... , spans=c(L, M, N), ... )
ベクトルで指定すると,3回修正ダニエルスムーザがかけられます.つまり,1回目は長さの修正ダニエルスムーザ,2回目は長さ
の修正ダニエルスムーザ,3回目は長さ
の修正ダニエルスムーザになります.
このように,複数回修正ダニエルスムーザをかけると,重みのバランスを変えられます.まだ,「確率密度関数の特性関数」と「畳込み定理」について説明していないので,ここからの説明は理解できない人がいるかもしれません.ポイントは,何回も何回も修正ダニエルスムーザ (あるいは,もとのダニエルスムーザ)をかけていくと,重み
が正規分布に近づきます.2回だと三角形に近い重み分布になります.

"spans"の設定により,平滑化をする重みがどのようになるのかを見るRスクリプトを載せておきます.
spans <- "c(5,5,5)"
w0 <- kernel("modified.daniell",m=eval(parse(text = spans)))$coef
w <- c(rev(w0[-1]),w0)
m <- (length(w)-1)/2
j <- (-m):(m)
w.max <- max(w)
plot(j,w,ylim=c(0,w.max*1.1),pch=16,col=2,las=1,yaxs="i",main=paste("spans =",spans))
arrows(j, 0, j, w, col =2,length=0,lwd=2)最初の行の
spans <- "c(5,5,5)"
の部分に,"spans=c(...)"で指定する右辺のベクトルを書いてください.

この記事も参考にしてください.
chaos-kiyono.hatenablog.com
おまけ
# 時系列の長さ (時系列は奇数長になります)
N <- 2^12
# 奇数に変換
N <- round(N/2)*2+1
# 半分
M <- (N-1)/2
#########################
# モデルの自己共分散関数を与える
# 例:AR(2)過程の自己共分散関数
AR2.model <- function(n,a1,a2,sig2){
Cov <- c()
Cov[1] <- sig2*(1-a2)/(1-a1^2-a2-a1^2*a2-a2^2+a2^3)
Cov[2] <- Cov[1]*a1/(1-a2)
for(k in 3:(n+1)){
Cov[k] <- a1*Cov[k-1] + a2*Cov[k-2]
}
return(Cov)
}
# パラメタの設定
a1 <- 1.6
a2 <- -0.9
sig2 <- 1
# 【注意】[-M,M]区間ではなく,[0,2M-1]にしている
acov.model <- c(AR2.model(M,a1,a2,sig2),rev(AR2.model(M,a1,a2,sig2)[-1]))
lag <- c(0:M,-(M:1))
#########################
# 自己共分散関数のフーリエ変換
fft.model <- fft(acov.model)
PSD.model <- Re(fft.model)
f <- c((0:M)/N,-(M:1)/(N))
#########################
# 白色ノイズの生成
WN <- rnorm(N)
# ホワイトノイズのフーリエ変換
fft.WN <- fft(WN)
#########################
# サンプル時系列の生成
fft.sim <- sqrt(PSD.model)*fft.WN
x.sim <- Re(fft(fft.sim,inverse=TRUE))/N
#########################
# 結果の描画
par(mfrow=c(2,3),cex.main=1.5,cex.axis=1.4,cex.lab=1.7,las=1,mar=c(5,5,2,2))
########################################################################
# PSDの推定
# spansなし
tmp <- spectrum(x.sim,plot="false")
freq <- tmp$freq
psd <- tmp$spec
#
plot(freq[freq > 0],psd[freq > 0],"l",log="xy",col=4,pch=16,xlim=c(f[2],0.5),xaxs="i",xlab="f",ylab="Periodogram",main=paste("without spans"),lwd=2,xaxt="n",yaxt="n")
lines(f[f > 0],PSD.model[f > 0],col=2,lwd=2,lty=2)
# 対数目盛を描く
axis(1,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(1,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
axis(2,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(2,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
########
legend("bottomleft", legend=c("Analytical Power Spectrum"), col=c(2), lty=c(2),lwd=c(2))
########################################################################
# PSDの推定
# spans=5
tmp <- spectrum(x.sim,spans=5,plot="false")
freq <- tmp$freq
psd <- tmp$spec
#
plot(freq[freq > 0],psd[freq > 0],"l",log="xy",col=4,pch=16,xlim=c(f[2],0.5),xaxs="i",xlab="f",ylab="Periodogram",main=paste("spans=5"),lwd=2,xaxt="n",yaxt="n")
lines(f[f > 0],PSD.model[f > 0],col=2,lwd=2,lty=2)
# 対数目盛を描く
axis(1,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(1,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
axis(2,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(2,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
########
legend("bottomleft", legend=c("Analytical Power Spectrum"), col=c(2), lty=c(2),lwd=c(2))
########################################################################
# PSDの推定
# spans=15
tmp <- spectrum(x.sim,spans=15,plot="false")
freq <- tmp$freq
psd <- tmp$spec
#
plot(freq[freq > 0],psd[freq > 0],"l",log="xy",col=4,pch=16,xlim=c(f[2],0.5),xaxs="i",xlab="f",ylab="Periodogram",main=paste("spans=15"),lwd=2,xaxt="n",yaxt="n")
lines(f[f > 0],PSD.model[f > 0],col=2,lwd=2,lty=2)
# 対数目盛を描く
axis(1,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(1,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
axis(2,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(2,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
########
legend("bottomleft", legend=c("Analytical Power Spectrum"), col=c(2), lty=c(2),lwd=c(2))
########################################################################
# PSDの推定
# spans=c(5,5)
tmp <- spectrum(x.sim,spans=c(5,5),plot="false")
freq <- tmp$freq
psd <- tmp$spec
#
plot(freq[freq > 0],psd[freq > 0],"l",log="xy",col=4,pch=16,xlim=c(f[2],0.5),xaxs="i",xlab="f",ylab="Periodogram",main=paste("spans=c(5,5)"),lwd=2,xaxt="n",yaxt="n")
lines(f[f > 0],PSD.model[f > 0],col=2,lwd=2,lty=2)
# 対数目盛を描く
axis(1,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(1,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
axis(2,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(2,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
########
legend("bottomleft", legend=c("Analytical Power Spectrum"), col=c(2), lty=c(2),lwd=c(2))
########################################################################
# PSDの推定
# spans=c(15,15)
tmp <- spectrum(x.sim,spans=c(15,15),plot="false")
freq <- tmp$freq
psd <- tmp$spec
#
plot(freq[freq > 0],psd[freq > 0],"l",log="xy",col=4,pch=16,xlim=c(f[2],0.5),xaxs="i",xlab="f",ylab="Periodogram",main=paste("spans=c(15,15)"),lwd=2,xaxt="n",yaxt="n")
lines(f[f > 0],PSD.model[f > 0],col=2,lwd=2,lty=2)
# 対数目盛を描く
axis(1,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(1,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
axis(2,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(2,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
########
legend("bottomleft", legend=c("Analytical Power Spectrum"), col=c(2), lty=c(2),lwd=c(2))
########################################################################
# PSDの推定
# spans=c(15,15,15,15,15,15)
tmp <- spectrum(x.sim,spans=c(15,15,15,15,15,15),plot="false")
freq <- tmp$freq
psd <- tmp$spec
#
plot(freq[freq > 0],psd[freq > 0],"l",log="xy",col=4,pch=16,xlim=c(f[2],0.5),xaxs="i",xlab="f",ylab="Periodogram",main=paste("spans=c(15,15,15,15,15,15)"),lwd=2,xaxt="n",yaxt="n")
lines(f[f > 0],PSD.model[f > 0],col=2,lwd=2,lty=2)
# 対数目盛を描く
axis(1,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(1,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
axis(2,at=10^(-5:5)%x%(1:9),label=FALSE,tck=-0.02)
tmp<-paste(paste(sep="","expression(10^",-5:5,")"),collapse=",")
v.label<-paste("axis(2,las=1,at=10^(-5:5),label=c(",tmp,"),tck=-0.03)",sep="")
eval(parse(text=v.label))
########
legend("bottomleft", legend=c("Analytical Power Spectrum"), col=c(2), lty=c(2),lwd=c(2))
##########################################################